【統計】生存時間解析 カプランマイヤー法(Kaplan-meier method)
カプランマイヤー法(Kaplan-meier method)についてのメモ。
【目次】
累積生存率とその信頼区間について記載する。
信頼区間はRで扱えるPlain(SASではLinear)、Log、Log-Logの3種類について。
計算式等
先に計算方法をまとめておく。
■ 累積生存率(Cumulative Survival Rate)
推定値
分散と95%信頼区間(Type:Linear(Plain))
※ここに示した分散はGreenwoodの公式と言われる。
分散と95%信頼区間(Type:Log)
分散と95%信頼区間(Type:Log-Log)
■ 生存期間中央値(Median Survival Time:MST)
※累積生存率が初めて 1 - p(MSTの時は50%)を下回る時点。
※p=0.5でMST。p=0.25, 0.75で25、75パーセンタイル値。
■ (参考)累積ハザード(cumulative hazard)
ノート
Kaplan-meier 法は、打ち切りデータ(Censored data)を考慮した上で生存率を計算する方法である。
生存時間解析というと死亡有無の評価に見えるが、関心イベントの発生を評価するため死亡以外にも使える(例:骨折、転倒など)。
打ち切りとは
関心イベントを追跡できなくなった状態を言う(※本記事では右側打ち切りのみ扱う)。
下図の例では、Bさんは観察期間内にイベントが発生せず打ち切り、Cさんは観察期間中に脱落で打ち切りとなる。
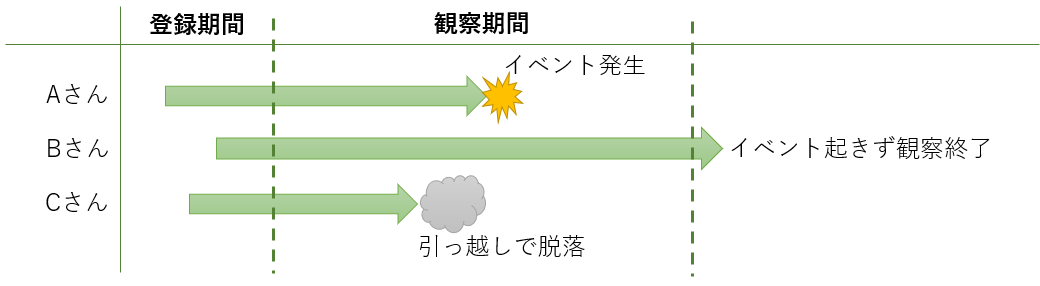
打ち切りデータは時点の後ろに「+」を付けて表し、例えば「10+」とすれば時点 t=10 での打ち切りとなる。
また、後述するKaplan-meier curveでは打ち切り時点に「+」をアノテーションする習わしがある(ヒゲと呼ばれる)。
サンプルデータ
今回は、RのMASSパッケージにあるgehanデータ(Remission Times of Leukaemia Patients)を用いる。
白血病患者の寛解の継続期間(週)に関するデータでイベントは再発。cens列に打ち切り情報(0=打ち切り、1=イベント)がある。Control vs. 6-MPの2群データであり、pairは群毎の連番を表す。
> library(MASS) > head(gehan, 10); pair time cens treat 1 1 1 1 control 2 1 10 1 6-MP 3 2 22 1 control 4 2 7 1 6-MP 5 3 3 1 control 6 3 32 0 6-MP 7 4 12 1 control 8 4 23 1 6-MP 9 5 8 1 control 10 5 22 1 6-MP
これをADTTEと名付けて、以降 RとSASで使用する。
ADTTE <- data.frame( AVAL = gehan$time ,EVENT = gehan$cens ,CNSR = ifelse(gehan$cens==0, 1, 0) #-- イベント=0, 打ち切り>0の形式 ,TRT01A = gehan$treat)
手計算してみる
6-MP群についてExcelで計算してみると下図の通りになる。

★Excel関数の中身(折りたたんでます。ここをクリック)
7列目: =IF(RC[-2] > 0,(RC[-3] - RC[-2]) / RC[-3], "") 8列目: =LN((RC[-5] - RC[-4]) / RC[-5]) 9列目: =IF(RC[-5] > 0, R[-1]C * RC[-3], R[-1]C) 10列目: =IF(RC[-4] <> "", (1 - RC[-4]) + R[-1]C, R[-1]C) 11列目: =RC[-2]^2 * RC[1] 12列目: =RC[-5] + R[-1]C 13列目: =RC[-5] + R[-1]C 14列目: =RC[-2] / RC[-1]^2 15列目: =RC[-6] - 1.96 * SQRT(RC[-4]) 16列目: =RC[-7] + 1.96 * SQRT(RC[-5]) 17列目: =EXP(LN(RC[-8]) - 1.96 * SQRT(RC[-5])) 18列目: =EXP(LN(RC[-9]) + 1.96 * SQRT(RC[-6])) 19列目: =EXP(-1 * EXP(LN(-1 * LN(RC[-10])) + 1.96 * SQRT(RC[-5]) )) 20列目: =EXP(-1 * EXP(LN(-1 * LN(RC[-11])) - 1.96 * SQRT(RC[-6]) ))
ここでは理解のため、6-MP群の3行目のデータまで式を出しながら計算してみる。
| 生存時間 (t) |
リスク集団 (n) |
イベント例 (d) |
打ち切り (c) |
生存率 1-d/n |
| 6 | 21 | 3 | 1 | 0.8571 |
| 7 | 17 | 1 | 0 | 0.9412 |
| 9 | 16 | 0 | 1 |
上表は生存時間解析を行う上での基本情報となる。
このうち、リスク集団は、前時点の「リスク集団 -(イベント例+打ち切り例)」から計算される(時点tになる直前のイベント未発生例)。
また、生存率は「1 -(イベント例÷リスク集団)」で計算し、打ち切り例は含めない。
累積生存率の推定値 S(t)
イベント発生時点を対象に当該時点までの生存率を掛け算するため、t=6、7は次の計算になる。また、t=9は打ち切りのみ(イベント未発生)のため、t=7の累積生存率のままとなる。
累積生存率の分散
まず Var[log S(t)] について例示する。
イベント発生時点を対象に「イベント例 / {リスク集団(リスク集団 - イベント例)} 」を足し算するため、t=6、7は次の計算になる。なお、t=9は打ち切りのみのため t=7と同じ。
次に Var[log (-log S(t))] について例示する。
Var[log S(t)] を「log{(リスク集団-イベント例)/リスク集団}」の累積和の二乗で割ることで求まるため、次のように計算できる。
最後に Var[S(t)] について例示する。
Var[log S(t)] に累積生存率S(t) の二乗を掛けることで求まるため、次のように計算できる。
累積生存率の95%信頼区間
信頼区間は上で求めた推定値・分散を用いるため、1行目の t=6 のみ例示する。
なお、信頼区間上限について、今回は例示のため1以上の数値も示したが、実際は0~1の区間を扱うため1以上は1とする。
生存期間中央値
MSTは累積生存率 S(t) が初めて0.5を下回った時点となる。
本サンプルでは S(22)=0.5378 、S(23)=0.4482 のため、MST=23となる。
Rでの実行
survivalパッケージとsurvminerパッケージを使ってKaplan-meier curveを描画する。
library(survival) library(survminer)
Kaplan-meier法はsurvivalパッケージのSurv関数とsurvfit関数を組み合わせて行う。
累積生存率の信頼区間(conf.type)はlog、log-log、plain を選択できるが、今回はlog-logで例示する。
KM1 <- survfit(formula = Surv(time=AVAL, event=EVENT) ~ TRT01A ,conf.type = "log-log" ,conf.int = .95 ,type = "kaplan-meier" ,error = "greenwood" ,data = ADTTE)
なお、Surv関数の部分では次のインプットデータを作っている。
「打ち切りとは」で触れたように数字の後ろに「+」が付くものが打ち切りデータである。
Surv(time=ADTTE$AVAL, event=ADTTE$EVENT) [1] 1 10 22 7 3 32+ 12 23 8 22 17 6 2 16 11 34+ 8 32+ 12 25+ 2 11+ [23] 5 20+ 4 19+ 15 6 8 17+ 23 35+ 5 6 11 13 4 9+ 1 6+ 8 10+
Kaplan-meier curveはSurv関数で取得した結果をsurvminerパッケージのggsurvplot関数に引き渡すときれいに描画してくれる。
ggsurvplot(KM1 ,pval = TRUE ,conf.int = TRUE ,risk.table = TRUE ,risk.table.col = "strata" ,linetype = "strata" ,surv.median.line = "hv" ,ggtheme = theme_bw() ,palette = c("blue", "darkgreen") )

なお、生存時間解析の分析データを集約すると次のとおり。
★データの中身(折りたたんでます。ここをクリック)
> summary(KM1) Call: survfit(formula = Surv(time = AVAL, event = EVENT) ~ TRT01A, data = ADTTE, error = "greenwood", conf.type = "log-log", conf.int = 0.95, type = "kaplan-meier") TRT01A=6-MP time n.risk n.event survival std.err lower 95% CI upper 95% CI 6 21 3 0.857 0.0764 0.620 0.952 7 17 1 0.807 0.0869 0.563 0.923 10 15 1 0.753 0.0963 0.503 0.889 13 12 1 0.690 0.1068 0.432 0.849 16 11 1 0.627 0.1141 0.368 0.805 22 7 1 0.538 0.1282 0.268 0.747 23 6 1 0.448 0.1346 0.188 0.680 TRT01A=control time n.risk n.event survival std.err lower 95% CI upper 95% CI 1 21 2 0.9048 0.0641 0.67005 0.975 2 19 2 0.8095 0.0857 0.56891 0.924 3 17 1 0.7619 0.0929 0.51939 0.893 4 16 2 0.6667 0.1029 0.42535 0.825 5 14 2 0.5714 0.1080 0.33798 0.749 8 12 4 0.3810 0.1060 0.18307 0.578 11 8 2 0.2857 0.0986 0.11656 0.482 12 6 2 0.1905 0.0857 0.05948 0.377 15 4 1 0.1429 0.0764 0.03566 0.321 17 3 1 0.0952 0.0641 0.01626 0.261 22 2 1 0.0476 0.0465 0.00332 0.197 23 1 1 0.0000 NaN NA NA > print(KM1) Call: survfit(formula = Surv(time = AVAL, event = EVENT) ~ TRT01A, data = ADTTE, error = "greenwood", conf.type = "log-log", conf.int = 0.95, type = "kaplan-meier") n events median 0.95LCL 0.95UCL TRT01A=6-MP 21 9 23 13 NA TRT01A=control 21 21 8 4 11
SASでの実行
Rの実行で使ったデータと同じものをLIFETESTプロシジャで実行する。
proc lifetest data=ADTTE plots=s(cl atrisk maxlen=30 outside) alpha=0.05 conftype=loglog;
time AVAL * CNSR(0);
strata TRT01A / test=logrank;
ods output SurvivalPlot = SP1;
ods output Quartiles = QT1;
run;
SASの場合は、LIFETESTプロシジャを実行すれば、累積生存率やグラフを一式出力してくれる。
グラフの設定はplotsオプションで行う。s : 累積生存率の描画、cl : 信頼区間の描画、atrisk maxlen=30 outside : グラフ外にリスク集団を表示。最大文字は30。
信頼区間はconftypeオプションで指定でき、loglog、log、linear等がある。デフォルトはloglog。また、alphaオプションで何%信頼区間にするか指定する。

ただし、実際はアウトプットしたデータセットを使って SGPLOTプロシジャで作り直すことが多いかもしれない。
累積生存率等は ods output SurvivalPlot で、MST等は ods output Quartiles でデータセット化できる。
プログラムコード
R
#-- Kaplan-meier method KM1 <- survfit(formula = Surv(time=AVAL, event=EVENT) ~ TRT01A #-- 1群の時はTRT01Aのところを 1 とする。 ,conf.type = "log-log" #-- log or log-log or plain ,conf.int = 0.95 ,type = "kaplan-meier" ,error = "greenwood" ,data = ADTTE) #-- Kaplan-meier curve ggsurvplot(KM1 ,pval = TRUE ,conf.int = TRUE ,risk.table = TRUE ,risk.table.col = "strata" ,linetype = "strata" ,surv.median.line = "hv" ,ggtheme = theme_bw() ,palette = c("blue", "darkgreen") )
proc lifetest data=ADTTE plots=s(cl atrisk maxlen=30 outside) alpha=0.05 conftype=loglog;
time AVAL * CNSR(0);
strata TRT01A / test=logrank;
ods output SurvivalPlot = SP1; /* 累積生存率などのデータセット化 */
ods output Quartiles = QT1; /* MSTなどのデータセット化 */
run;
整備中
参考
統計学(出版:東京図書), 日本統計学会編
https://support.sas.com/documentation/onlinedoc/stat/142/lifetest.pdf
http://www.math.ucsd.edu/~rxu/math284/slect2.pdf
https://www.pharmasug.org/proceedings/2014/SP/PharmaSUG-2014-SP10.pdf
https://www.sas.com/content/dam/SAS/ja_jp/doc/event/sas-user-groups/usergroups11-a-02.pdf